Bayesiansk fylogenetisk analys
Sammanfattning
Bayesiansk statistik har fått sitt namn efter Thomas Bayes, som var en präst och amatörmatematiker som levde på 1700-talet. Han visade hur man ”vänder” på sannolikheter, så att man kan räkna ut sannolikheten för att en hypotes är korrekt. I vanlig statistik måste man angripa den frågeställningen indirekt.
Bayes metod ledde till svåra matematiska ekvationer som inte kunde lösas med papper och penna. På 1950-talet utvecklade emellertid forskare i det amerikanska kärnvapenprogrammet effektiva numeriska metoder för att lösa dessa problem, och i kombination med dagens kraftfulla datorer har det gjort Bayesiansk statistik till en standardmetod inom de flesta vetenskapsområden.
Vi var bland de första som utvecklade program och algoritmer för Bayesiansk inferens av evolutionära släktskapsträd. Idag rekonstrueras evolutionära träd – eller fylogenier som de också kallas – till stor del från genetiska data. Användningsområdena för fylogenetisk analys är förvånansvärt många. Den kan användas för att spåra smittvägar vid utbrott av virussjukdomar, ta reda på när och var olika grupper uppstod, och lista ut hur olika språk uppstod ur varandra, bara för att nämna några exempel.
Vårt arbete sker till stor del inom ramen för programmen MrBayes och RevBayes. Vi intresserar oss bland annat för hur evolutionära modeller kan formuleras på ett generiskt sätt, och vi arbetar med att förbättra de numeriska metoderna som används. Vi arbetar också med flera olika tillämpningar inom evolutionsbiologin, exempelvis när det gäller att förstå hur morfologiska karaktärer förändras över tid och hur fossil kan användas för att datera evolutionära händelser.
Länkar
MrBayes (http://mrbayes.net )
)
RevBayes (http://revbayes.com )
)
Forskningen stöds av Vetenskapsrådet
Projektdeltagare vid NRM
Fredrik Ronquist (Professor, forskningsledare)
Allison Hsiang (Postdoktor)
Johan Nylander (Bioinformatiker)
Utvalda publikationer
Ronquist F, Teslenko M, van der Mark P, Ayres DL, Darling A, Höhna S, Larget B, Liu L, Suchard M, Huelsenbeck JP. 2012. MrBayes 3.2: Efficient Bayesian phylogenetic inference and model choice across a large model space. Systematic Biology 61: 539–542.
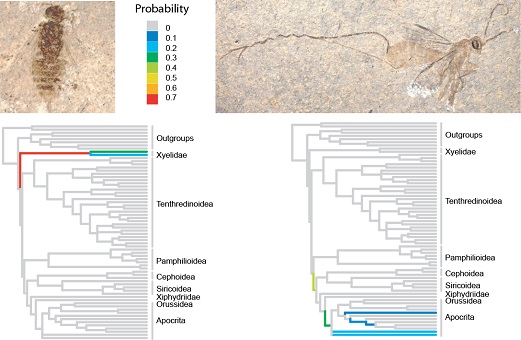
Ronquist F, Klopfstein S, Vilhelmsen L, Schulmeister S, Murray D, Rasnitsyn AP. 2012. A total-evidence approach to dating with fossils, applied to the early radiation of Hymenoptera. Systematic Biology 61: 973–999.
Höhna S, Heath T, Boussau B, Landis M, Ronquist F, Huelsenbeck JP. 2014. Probabilistic graphical model representation in phylogenetics. Systematic Biology 63: 753-771.
Klopfstein S, Vilhelmsen L, Ronquist F. 2015. A nonstationary Markov model detects directional evolution in hymenopteran morphology. Systematic Biology 64: 1089–1103.
Zhang C, Stadler T, Klopfstein S, Heath TA, Ronquist F. 2016. Total-Evidence Dating under the Fossilized Birth-Death Process. Systematic Biology 65: 228–249.
Ronquist F, Lartillot N, Phillips MJ. 2016. Closing the gap between rocks and clocks using total-evidence dating. Philosophical Transactions of the Royal Society of London B 371: 20150136.
Lartillot N, Ronquist F, Phillips MJ. 2016. A mixed relaxed clock. Philosophical Transactions of the Royal Society of London B 371: 20150132.
Höhna S, Landis MJ, Heath TA, Boussau B, Lartillot N, Moore BR, Huelsenbeck JP, Ronquist F. 2016. RevBayes: Bayesian Phylogenetic Inference Using Graphical Models and an Interactive Model-Specification Language. Systematic Biology 65: 726–736.